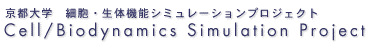分散計算による感度解析の高速化
はじめに
これまでも物理学や工学分野では観測された現象を数理モデルで表現して理解するのが当たり前であったが、 我々は心臓機能の包括的な数理モデルを開発し、仮説駆動型医学研究を進めている(Takeuchi et al, 2006)。 すなわち、心筋細胞を構成する個々の機能要素を数式としてモデル化し、 その相関系としての細胞機能を数理モデルとして表現する。 そしてシミュレーションを繰り返し、モデルそのものと実験結果を検証し、可視化することが新たな仮説につながっている(図1)。
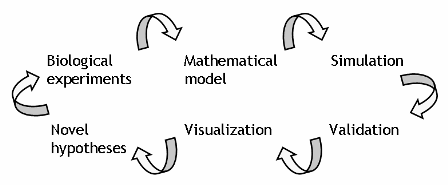
Fig. 1. Image of model-driven biological study. Mathematical modeling is common in engineering field, and will be a great accelerator for medical and biological study.
このとき、モデルを構成するパラメータそれぞれについて、モデル全体の振る舞いに及ぼす影響を解析する必要があるにもかかわらず、 高度な計算機能力が必要であることからこれまでそのような解析は数学者の手に委ねられていた。
我々の研究室では、広く利用されており、 学びやすいJava (http://www.java.com/ja/) を用いて微分方程式を記述し、XML (http://www.xmlconsortium.org/) 形式でパラメータの初期値と、機能要素の組み合わせを記載するシミュレータ、 simBio (http://jp.sim-bio.org/)を開発し、 数理モデルの開発および解析に利用している。 そこには生物系研究者自身の手で感度解析を実施でき、解析法を記録・再利用可能とし、速やかに繰り返しうるシステムが必要である。
別個のパラメータが設定されたモデル同士は相互に独立して計算しうるので、クラスタを用いた分散計算が適している。 そこでネットワークに接続された複数のPCを計算資源として活用する分散計算を利用し、 任意の数のパラメータを同時に変更して、ある評価値が示す依存性を速やかにフィードバックする感度解析システムを開発したところ、 大きな効果が得られたのでここに報告する。
方法
Javaで利用可能な分散計算用パッケージとしてオープンソースで開発されている Jini (http://www.jini.org/) を利用した。 JiniのサービスであるJavaSpacesはネットワーク上に共有メモリを提供する仕組みである。 そこで、この共有メモリ上に計算要求としてモデルxmlが書き込まれたらそれを取り出し、 simBioで計算を実行し、結果xmlを共有メモリに書き込む計算サーバー(ワーカー)を開発した。 生体モデルに様々なパラメータセットを設定したxmlを計算要求として書き込み、 計算結果xmlが戻ってきたら、それを取り出して集計するクライアントを開発した。 もう一つ、ワーカーを停止させるクラスの合計3クラスを開発し、 JavaSpacesサービスの起動と登録にはサンプルコードを利用した。
simBioは各研究者が独自に開発したモデルを相互に自由に結合できるところにその特色がある。 従って、開発過程では各研究者は共通部分と独自開発した部分とを結合したモデルを作成している。 すなわち、各研究者が計算に利用したいJavaクラスをワーカーが認識する必要がある。 これを実現するため計算要求xmlに特定のクライアントの持つJavaクラスを使う指定を対応させた。 Javaクラスをワーカーに受け渡すには、Javaで標準的に用いられるhttpクラスサーバーを利用した。
これにより、システムでは以下のようなメッセージが受け渡され分散計算が実行される。
- クライアントがモデルxmlをJavaSpacesに書き込む。
- 起動され、待機していたワーカーがモデルxmlをJavaSpacesから読み込む。
- ワーカーが所持しているJavaクラスがモデルxmlを計算するためのJavaクラスそのものの最新版であるか、httpクラスサーバーに確認する。
- ワーカーが所持しているJavaクラスが最新版でなければ、最新版をhttpクラスサーバーから受け取る。
- ワーカーがsimBioを使ってモデルxmlの計算を開始する。
- ワーカーがモデルxmlの計算開始をJavaSpacesに書き込む。
- クライアントがモデルxmlの計算開始をJavaSpacesから読み込み、次の計算要求を書き込む。
- simBioの計算が終わったら、ワーカーが結果xmlを書き込む。
- クライアントが結果xmlをJavaSpacesから読み込み、領収証を書き込む。
- ワーカーが領収証を読み込んだら控えの結果xmlを破棄する。
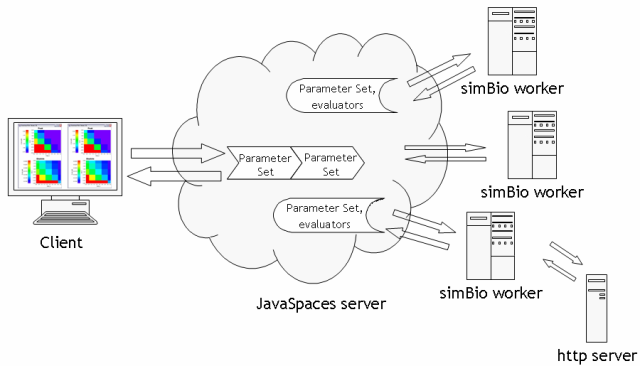
Fig. 2. Schematic diagram of distributed computing. Client, and each server of Javaspaces, simBio, and http can be run on separate computers or on the same computer.
研究者に速やかなフィードバックを行うために、計算終了と同時に集計結果を可視化する機能を実装した。 変化させたパラメータが2個の場合は2次元カラーマップで結果を表示した。 同時に、他のソフトを用いて可視化できるように、 変更したパラメータ値と評価値をテーブルにした単一のテキストファイルとしても結果集計表を出力した。
我々の研究室ネットワークには種々のPCが混在している。 Hyper Thread機能を搭載したCPUについてはそれを有効にし、複数のモデルを同時に計算した。 RAMは最低1GBを搭載していた。 Java VMはIBM JDK 1.4.2を用いた。OSはX1についてはRed Hat Linuxを、それ以外はWindows XPを用いた。
分散計算を用いた感度解析は以下の手順で実行出来るようシステム化した。
- クライアントだけを用いて、感度解析用xmlの動作を確認する。このとき計算時間を短く、回数を少なくしても良い。
- JavaSpacesサーバーを起動。
- ワーカーを起動。
- httpクラスサーバーを起動。
- クライアントで分散計算の利用を指定して感度解析用xmlを実行。
- 可視化された結果を解釈。
結果
2つのパラメータNCXとPMCAを同時に変化させたとき、評価値Caiの最大値と最小値の振る舞いを図3に示す。 図3Aは上記手順1で得られた結果であり、図3Bは手順6であり、論文(Sarai et al, 2006)に用いた図と同じである。 共に計算直後に解析結果が可視化された。 同じ評価値が得られるパラメータの組み合わせが無数に存在していることが一目瞭然である。 分散計算を利用することで、同じ待ち時間で、図3Aの代わりに図3Bが得られた。
Fig. 3. Two-dimensional color contour plot by a single PC with 2 Xeon CPUs (A) and by 11 PCs (B). Figure B was modified from Fig. 3B in Sarai et al. (2006).
図3Bを構成する21x24 = 合計504通りのパラメータセットについて、 評価値を得るのに要した実時間(total)と、 PC 1台で計算させた場合の予想所用時間を図4に示す。 1つのパラメータセット毎に定常状態に達するまで計算を行った。 11台のPC、計22スレッドを用いたところ、6時間1分で全ての計算が終了した。 Pentium 4 2.6 GHz Hyper Thread単独(P4, P5に相当)の場合と比較すると、 予想所用78時間に対して約13分の1に短縮された。
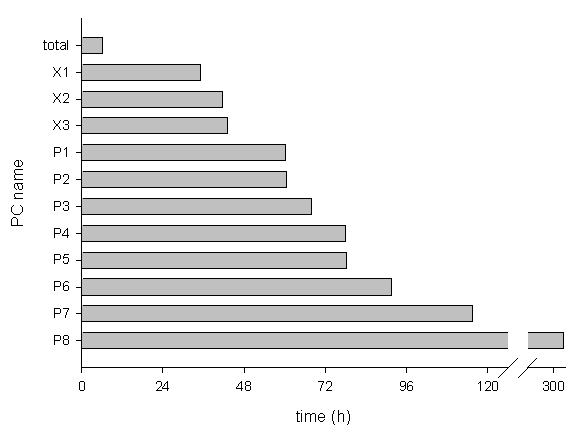
Fig. 4. Elapsed time of computing 504 models by 11 PCs vs estimated time by each PC. PCs with Pentium CPU were labeled as P1 to P8, and PCs with dual Xeon CPUs were labeled as X1 to X3. Estimated time by each PC was extrapolated by the number of calculated models in the elapsed time.
考察
数理モデルが複雑化するにつれ、その振る舞いを理解し、検証するために要する時間が莫大なものになりつつある。 パラメータの変更による振る舞いの変化を速やかに可視化することはモデルを用いた研究に不可欠である。 今回作成した分散計算システムは以下の点で有用であると考えられる。
- 解析結果がすぐに可視化される。
- 1台のPCを利用するのも、分散計算を利用するのも同じ手順ファイルをそのまま利用できる。
- 分散計算を利用することで、同じ待ち時間でより詳細な結果を得られる。
- 解析手順が記録されているので、その再現や改変が自由に出来る。
- 数理モデルそのものを作成・解析した手順全体をそのまま記録できる。
今回、オープンソースプロダクトを利用することで、必要最小限のプログラム、すなわちJavaクラスとして3クラスを記述するだけで、分散計算を活用できた。 様々なパラメータセットを与えて、計算するというジョブは粒度が大きく、 通信が少ない問題であるため、それぞれのPCの処理時間に応じて分散の効果が現れた。 そのため、利用する計算機資源を追加することで、直線的にその効果、 すなわち結果が集計されるまでに要する時間が短縮された。 今回の手法は、通常の生物学研究室において、数理モデルを利用する上で、 既にある計算機資源を有効に活用でき、 さらに、漸増的に計算機資源を追加することでその効果を期待できる有効な手法である。
利用するシステムが高度化すればするほど、不確実性と管理コストが増加する。 感度解析を実行する手順は6ステップまで単純化できたが、より簡単に利用できるよう整備する必要がある。 今回のシステムはJavaとJavaSpacesの提供する機能に多くを依存しているが、JavaSpacesは大量にメモリを消費した。 JavaSpacesサーバーがメモリ不足で停止する危険性、通信過程で計算結果を失う危険性に対処するため、 ワーカーに控えを保存する必要があった。 複数のクライアントからの計算要求の効率的な処理や、研究室内に限られないネットワークや、 確実性がより要求される環境での利用などに対応するためには、 商用に改良されたJavaSpacesの利用や、より堅牢な分散計算環境への移植なども考慮する必要がある。
今回、我々はオープンソースの成果とクラスタを用いて、 パラメータ値に応じてどのように数理モデルの振る舞いが変化するかを、短時間で可視化できた。 著者は、分散計算を利用するプログラムの作成に3日を要したが、その投資は十二分に回収されている。
感度解析システムはwww.sim-bio.orgから自由に入手して利用可能である。
参考文献
- Susanne Hupfer. JavaSpacesプログラムの中に分散データ構造を構築して使用する方法 [Online]. Java technology in developerWorks, IBM, 2000. http://www-06.ibm.com/jp/developerworks/java/010323/j_j-space.html [14 Nov. 2005].
- Korpela, Eric, Werthimer, Dan, Anderson, David, Cobb, Jeff, Lebofsky, Matt, SETI@home - Massively Distributed Computing for SETI , Computing in Science & Engineering, 78-83, 2001.
- N Sarai. Acceleration of sensitivity analysis by distributed computation. Conf Proc IEEE Eng Med Biol Soc 1:1144-7, 2007.
- N Sarai, T Kobayashi, S Matsuoka and A Noma. A Simulation Study to Rescue the Na+/Ca2+ Exchanger Knockout Mice. J Physiol Sci 56(3): 211-217, 2006. doi:10.2170/physiolsci.RP005006
- N Sarai, S Matsuoka and A Noma. simBio: a Java package for the development of detailed cell models. Progress in Biophysics and Molecular Biology, 90(1-3): 360-377, 2006.. [The software is available at http://www.sim-bio.org/]
- A Takeuchi, S Tatsumi, N Sarai, K Terashima, S Matsuoka, and A Noma. Ionic Mechanisms of Cardiac Cell Swelling Induced by Blocking Na+/K+ Pump As Revealed by Experiments and Simulation. J Gen Physiol 128(5): 495-507, 2006. doi:10.1085/jgp.200609646
- 皿井 伸明、天野 晃、松岡 達、松田 哲也、野間 昭典. 生物学的視点に基づくオブジェクト指向生体機能シミュレーション. シミュレーション, vol. 23, No. 1, p. 4-13, 2004. PDF (738 kb) [ソフトウェアはhttp://jp.sim-bio.org/から入手できます]
連絡先
皿井伸明
Eメール: nsarai _at_ users.sourceforge.net